Our Vision and Research
私たちのビジョンと研究
我们的研究构想和方向

Starting from a fertilized egg, cells proliferate, pass their genomic information to their offspring, and dynamically change their functions to form diverse tissue structures. Throughout development, intracellular and environmental cues trigger patterns of gene expression that govern cell state transitions and produce additional cellular and environmental cues, leading cells to self-organize into functional clusters within spatially distinct areas. How can these processes be investigated? High-resolution molecular snapshots of cells can be obtained using various omics technologies, but these methods require the destruction of the sample, thereby precluding time-course analyses. Live-cell imaging with fluorescent probes can analyze time-course dynamics but this method is limited to analyzing only a small number of molecules or cells. To overcome this common obstacle in biology, our research program is pioneering two major fileds in biology: DNA event recording and retrospective clone isolation.
Keywords:
Synthetic Biology, Computational Biology, Developmental Biology, Stem Cell Biology, Mouse Engineering, High-Performance Computing

動物の発生は受精卵からはじまり、細胞が分裂を繰り返すことで成立します。この過程で、細胞は遺伝情報を娘細胞に伝え、さまざまな組織構造を形成するためにその機能を動的に変化させます。また、環境シグナルはゲノムプログラムを通じて細胞内の遺伝子発現を変化させ、それらが次の細胞の内部状態を規定し、次のシグナルを外部環境のために作り上げます。このことが細胞が組織の異なる空間で機能的なクラスターに自己組織化することを実現させています。このような美しいプロセスはどのように調べることができるでしょうか?近年のオミクスプロファイルを計測する手法はいずれもサンプルの破壊を必要とするため、時系列分析を行うことができません。蛍光プローブを用いたライブセルイメージングなどは、少ない数の分子や細胞を対象とする観察に限ります。生物学には、哺乳動物個体などの多細胞から構成される複雑なシステムのダイナミクスを観測できないという大きなハードルがあります。これを克服するために、UBCおよび大阪大学にある私たちの研究室では、DNAイベントレコーディングと遡及的クローン単離という新しい生物学の2領域を提案しています。
キーワード:
合成生物学, 生命情報科学, 発生生物学, 幹細胞生物学, マウス工学, 大規模コンピューティング

动物的个体发育始于一枚受精卵。受精卵不断分裂,将其基因组信息传递给子细胞,同时改变自身功能以形成各种组织结构。细胞功能和状态的转换由环境信号所激发的基因表达所调控。由此,细胞产生新的分子信号,诱导它们在空间上的不同区域构建出功能各异的细胞群。我们该如何揭开这一高度复杂生命现象的神秘面纱?利用近年飞速发展的多组学分析技术,我们可以获得细胞的高分辨率分子快照。然而,这些方法需要破坏样本,因此无法实现时间序列上的分子动态追踪。此外,使用荧光探针进行活细胞成像也仅限于少量分子或细胞的实时观测。为了突破这些技术上的难关,我们在加拿大不列颠哥伦比亚大学和日本大阪大学的研究团队正共同开拓两个全新的生物学领域,即DNA事件记录 (DNA event recording) 和回溯性克隆分离 (Retrospective clone Isolation)。
关键词:
合成生物学,生物信息学,发育生物学,干细胞生物学,小鼠基因工程,大规模计算
DNA event recording
DNAイベントレコーディング
DNA事件记录
In the idea of DNA event recording, molecular and cellular events of a multicellular organism are progressively stored in synthetic “DNA tapes,” like a molecular ticker tape (Science 2022). Such a system allows for the readout of historical molecular expression profiles of many cells using high-throughput single-cell sequencing. Analogous to a video camera system, the requirements of DNA event recording systems are: (1) high-capacity DNA “memory” modules embedded in chromosomes; (2) highly sensitive “sensor” modules to capture cell divisions and cell types; (3) information “writer” modules to alter DNA tapes; and (4) high-capacity information “reader” modules to reconstruct complex biological history information written in DNA tapes.
DNAイベントレコーディングでは、複雑な生命システムを構成する細胞それぞれが、経験するイベントを細胞内に搭載されるビデオカメラをもちいて観察し、人工的なDNAのテープにに逐次的に記録します (Science 2022)。このようなシステムを実現することができれば、システムのライフコースを通じて細胞が経験する様々な情報を1細胞シークエンシング技術を利用して読み出し、データマイニングによって再構成できると考えています。DNAイベントレコーディングは、ビデオカメラのアナロジーそのままに、(1) 染色体に埋め込まれた高容量DNA「メモリ」モジュール、(2) 細胞分裂や細胞状態遷移などを捉える高感度「センサー」モジュール、(3) 観測されたイベントに応じてDNAテープを編集するための情報「書込み」モジュール、および (4) DNAテープに書かれた複雑な生物学的ヒストリー情報を再構築するための「読出し」モジュールの開発が必要になります。
在DNA事件记录系统中,搭载于细胞内的“摄像机”观测复杂生命系统中每个细胞中的分子动态,并将它们按顺序记录在“人造DNA磁带”中 (Science 2022)。通过这个系统,我们可以利用高通量单细胞测序技术读出细胞在生命过程中所经历的各种事件,并通过数据挖掘技术来进行重构。DNA 事件记录系统与摄像机类似,由以下部分组成: (1) 嵌入染色体中、作为信息存储介质的高容量DNA存储器 (memory),(2) 由合成基因回路所构成、监测细胞分裂和状态转换的高灵敏度传感器 (sensor),(3) 根据观测到的事件编辑 DNA 磁带的信息写入装置 (writer),以及 (4) 用于重建记录在 DNA 磁带中的复杂生物历史信息的事件读取模块 (reader)。
Molecular recorders to track cellular events.
Retrospective Clone Isolation
遡及的クローン単離
回溯性克隆分离
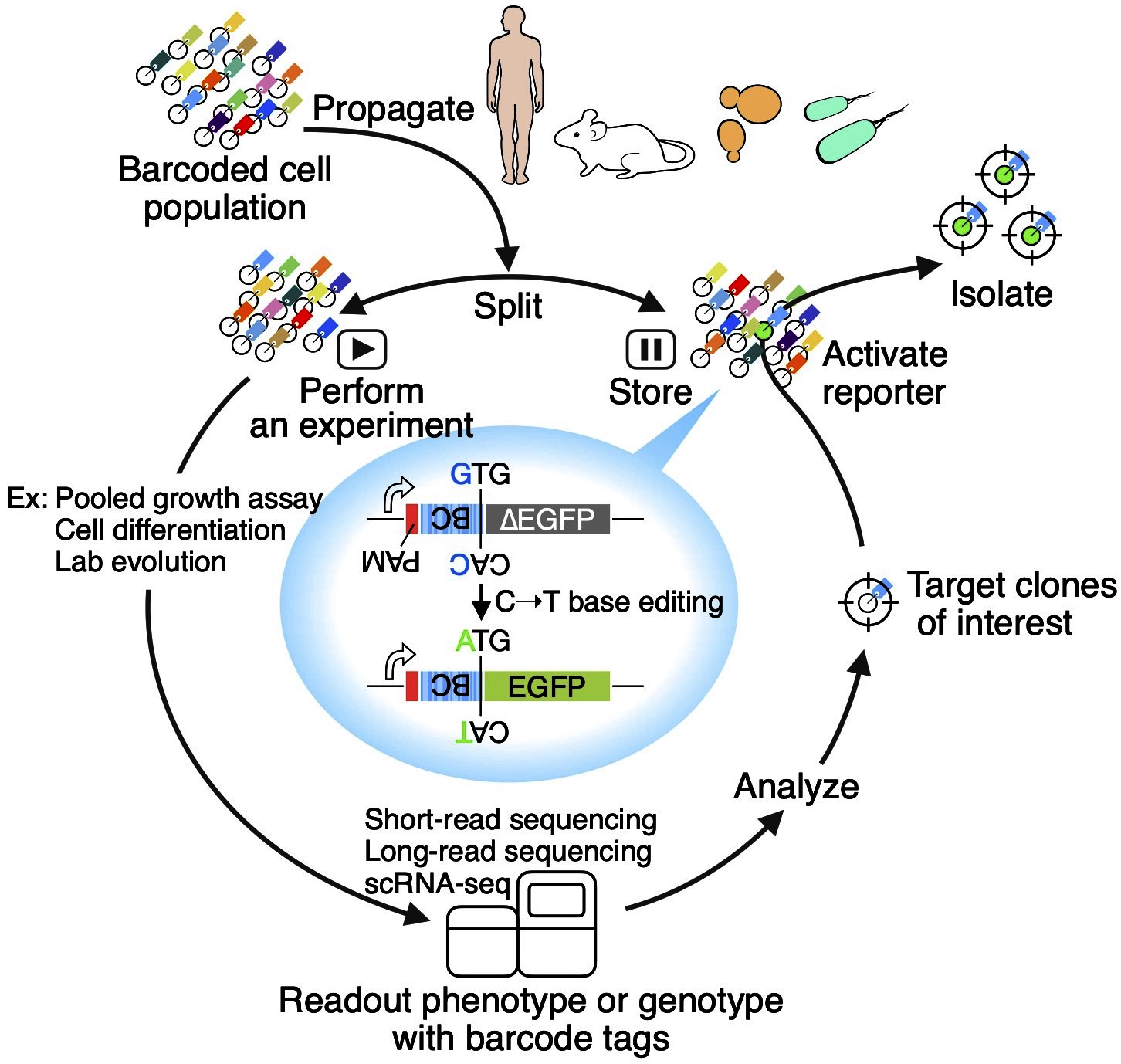
Another idea to achieve dynamic, high-content observation of progressive systems is retrospective clone isolation. In this concept, cells in a population are first tagged with unique, short DNA barcodes and propagated. A subpopulation is then subjected to a specific assay. After identifying a barcode for a clone of interest, the same clone, or its close relatives, is isolated in a barcode-dependent manner from the initial population or any other subpopulation stored during the experiment. The isolated clone can be subjected to various experiments, including omics measurements and synthetic population reconstitution assays. We recently established a multi-kingdom, highly efficient retrospective clone isolation method using CRISPR base editing (bioRxiv 2022). We are currently working on deciphering the molecular priming factors that determine cell fates in different human pluripotent stem cell differentiation and organoid models.

複雑に進展する多細胞システムを高解像で観察するためのもう一つのアイディアは、細胞をタイムマシーンのように遡って観察する追溯的クローン単離です。このアイディアでは、まず、集団内の細胞をそれぞれ特異的な短いDNAバーコードでタグ付けします。これをわずかに増殖させたあと、その一部(サブポピュレーション)を目的のアッセイにかけます(幹細胞の細胞分化、オルガノイド構築、がん細胞の増殖実験など)。アッセイのあとにあるDNAバーコードをもっているクローンが優勢的に特定の形質を示す細胞となった場合は、これと同じクローンを、初期に保存されたサブポピュレーションからバーコード依存的に単離します。単離したクローンは、さまざまなオミクス計測や運命の特定されたクローンの複合による人工的な細胞社会の再構成などに使用することができます。私たちは最近、CRISPR塩基編集を利用した高感度の追溯的クローン単離手法CloneSelectを確立しました (bioRxiv 2022)。現在、この手法をもちいて、さまざまなヒト多能性幹細胞の分化およびオルガノイド構築プロセスにおいて初期状態の細胞の分化運命を決定するプライミング因子を同定しようとしています。

另一种对多细胞生物系统中复杂细胞过程的高分辨率观测的方法是回溯性克隆分离。首先,我们使用特定的DNA条形码分别标记初始细胞群中的每个细胞,并将它们用于细胞生长实验,例如干细胞分化、类器官培养、以及癌细胞增殖。在对细胞亚群进行观察和分析过程中,我们将确定感兴趣的细胞克隆,并识别它们所携带的DNA条形码。采用针对该条形码的报告系统,我们将从初始细胞群或实验期间储存的任何细胞亚群中提取出相同的克隆或其近亲。分离出的克隆可用于多组学分析,或通过结合特定命运的克隆进行细胞种群的合成重组。最近,我们开发了一种基于CRISPR 碱基编辑技术的多物种、高灵敏度的回溯性克隆分离技术CloneSelect (bioRxiv 2022)。利用该方法,我们正致力于破译在各种人类多能干细胞分化和类器官模型中决定细胞分化命运的启动因子。
A multi-kingdom genetic barcoding system for precise clone isolation.
Nature Biotechnology 2025 PubMed PDF
+Equally contributed
Contribution to Specific Fields
これまでの成果
迄今取得的成果
To achieve these visions, we have incrementally worked on diverse topics in biology.
上記のビジョンを達成するために、私たちは生物学の様々な分野において研究開発を行っています。
为了实现上述研究构想,我们正在生物学的各个领域开展研究和开发工作。
Genome editing
ゲノム編集
基因组编辑
In CRISPR-Cas9 genome editing, a guide RNA recruits Cas9 to a target genomic region adjacent to a protospacer adjacent motif (PAM) sequence, 5’-NGG-3’, and Cas9 produces a DNA double-stranded break (DSB). This event promotes either gene deletion or transgene insertion through the induction of different DNA repair pathways, but DSBs are cytotoxic and the DSB-based editing outcomes are unpredictable. In collaboration with Dr. Keiji Nishida, we developed a new genome-editing tool, Target-AID, which fused cytidine deaminase (AID) to a nickase Cas9 and enabled highly precise targeted C→T substitutions without DSBs (Science 2016). In collaboration with Dr. Osamu Nureki, we also succeeded in increasing the targeting scope of Cas9 from a restrictive NGG to an NG PAM (Cas9-NG) and developed Target-AID-NG (Science 2018). Furthermore, my team developed a new base editor Target-ACEmax, which enables the simultaneous induction of C→T and A→G substitutions on a target DNA molecule and greatly expands the potential of base editing in therapeutics and biotechnology development (Nature Biotechnology 2020). Furthermore, to explore new genome editing tools apart from the CRIPSR-Cas9 and other characterized genome editing tools, we have developed a tool to rapidly capture periodic and interspaced periodic repeats from genomic and metagenomic resources (Nucleic Acids Research 2019).

CRISPR-Cas9ゲノム編集では、PAMと呼ばれるNGG配列に隣接するゲノムのターゲット配列と相補鎖を形成するガイドRNAがCas9をリクルートし、DNA二本鎖切断を生じさせます。これを利用して、遺伝子の欠失や外来遺伝子の効率的なノックインが実現できますが、DNA二本鎖切断には細胞毒性がある上に副産物として様々な予測できない編集結果ももたらします。私たちは神戸大学の近藤昭彦先生や西田敬二先生との共同研究により、2016年に第一世代のCRISPR塩基編集技術を開発しました (Science 2016)。これは、シチジン脱アミノ化酵素をニッカーゼ型のCas9に融合させたもので、DNA二本鎖切断なしで高精度に標的とするCをTに置換できる技術となりました。また、2018年に東京大学の濡木理先生と西増弘志先生がCas9のPAMを制限的なNGGからNGに拡張した際には、Target-AID-NGを開発しました (Science 2018)。さらに、2020年には新しい塩基編集ツールTarget-ACEmaxを開発しました (Nature Biotechnology 2020)。これは、標的DNA分子上でC→TおよびA→Gの異種塩基同時置換を可能にし、治療薬およびバイオテクノロジー開発における塩基編集の可能性を大きく拡張しました。さらに、CRISPR-Cas9や他のゲノム編集ツールとは別の新しいゲノム編集ツールを探究するために、ゲノムおよびメタゲノムリソースから周期的リピートを高速に探査するソフトウェアも開発しました (Nucleic Acids Research 2019)。

在CRISPR-Cas9基因组编辑系统中,向导 RNA (gRNA) 通过碱基互补配对识别邻近NGG (PAM序列) 的目标序列并引导Cas9蛋白产生DNA双链断裂。该系统可用于高效DNA片段的敲入和敲除,但DNA的双链断裂具有细胞毒性,并会产生不可预测的基因编辑副产品。2016年,我们与神户大学的Akihiko Kondo和Keiji Nishida博士合作开发了第一代CRISPR碱基编辑器Target-AID (Science 2016)。 这项技术通过DNA脱氨酶与Cas9缺口酶的融合,在不造成DNA双链断裂的同时将碱基C精确地替换为T。2018年,我们和东京大学的Osamu Nureki和Hiroshi Nishimasu博士将Cas9的PAM序列从NGG拓展为NG,并开发了识别NG的Target-AID (Science 2018)。2020 年,我们制作出全新的碱基编辑工具Target-ACEmax (Nature Biotechnology 2020),实现了在目标序列上同时进行C→T和A→G碱基置换,极大扩展了碱基编辑在基因治疗和生物技术领域中的应用前景。此外,我们还打造了从基因组和宏基因组资源中快速探测周期性重复序列的软件,以此探索除CRISPR-Cas9及其他基因编辑器以外的新型基因组编辑工具 (Nucleic Acids Research 2019)。
Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems.
Engineered CRISPR-Cas9 nuclease with expanded targeting space.
Base editors for simultaneous introduction of C-to-T and A-to-G mutations.
Nature Biotechnology 38, 865-869, 2020 PubMed PDF
+Equally contributed
Fast and global detection of periodic sequence repeats in large genomic resources.
Nucleic Acids Research 47, e8, 2019 PubMed PDF
Cell lineage tracing
細胞系譜トレーシング
细胞谱系追踪
Several methods have been proposed to trace developmental cell lineages of multicellular organisms, whereby chromosome-embedded DNA barcodes are continuously mutated by Cas9 and inherited from mother to daughter cells, whose lineage can be reconstructed according to the mutation patterns at the time of observation. However, none of these technologies has achieved high-resolution lineage tracing. To decipher the map of the whole-body mammalian (mouse) developmental process at the single-cell resolution, we have outlined the key issues and perspectives in this field (Science 2022) and are developing new genetic circuits, mouse engineering, and high-performance computing technologies. Target-AID and Target-ACEmax, described above, were developed primarily for the purpose of high-resolution cell lineage tracing. We also developed a new deep distributed computing platform and succeeded in the precise lineage reconstruction of over 235 million mutated sequences generated by a simulator (Nature Biotechnology 2022).
DNAイベントレコーディングのなかでも、発生における細胞系譜を記録して追跡する手法が先行して開発されています。特に、CRISPR-Cas9ゲノム編集をもちいる手法では、細胞の染色体に組み込まれたDNAバーコードがCas9によって連続的に変化し、母細胞から娘細胞へと受け継がれます。このシステムによって生まれた娘細胞群は、それらの持つ変異パターンを観測時にDNAシークエンシングによって同定することができるので、(変異パターンから進化系統樹を構築するようになデータ解析によって)細胞系譜を再構築することができます。しかし、これまでに開発されたいずれの技術も高解像度の細胞系統トレーシングを達成できていません。私たちは1細胞分裂解像度で哺乳類(マウス)全身の発生プロセスを解読するために必要な課題とビジョンを明らかにして (Science 2022) 戦略的に研究プロジェクトを展開しています。これには、新しい遺伝子回路、マウス工学、および大規模コンピューティング技術の開発などが含まれます。上記のTarget-AIDおよびTarget-ACEmaxも、高解像度の細胞系譜トレーシングのために開発されました。また、新しい深層分散コンピューティング技術を開発して、2億以上の配列から成る超巨大な系統樹を再構成する技術も樹立しています (Nature Biotechnology 2022)。
近年,研究者开发出了基于基因组编辑系统的细胞谱系追踪法,即使用CRISPR-Cas9系统编辑嵌入细胞染色体中的DNA条形码。经过细胞分裂,变异的条形码由母细胞传递给子细胞,并在子细胞中发生进一步的突变。随后,子细胞群所携带的变异条形码由DNA测序读出,并通过序列分析来重构细胞谱系 (类似于根据DNA的突变模式来构建进化系统树)。然而,这些技术仍无法实现高分辨率的细胞谱系追踪。为实现以单次细胞分裂的分辨率破译哺乳动物 (小鼠) 全身发育过程的详细图谱,我们概述了这一领域的关键问题和前景 (Science 2022),并致力于开发新的合成基因回路、基因工程小鼠、以及高性能计算技术。上文提及的碱基编辑工具Target-AID和Target-ACEmax主要是为高分辨率细胞谱系追踪而开发的。另外,我们还开发了一个新的深层分布式计算框架,成功对模拟器生成的超过2.35亿个突变序列进行了精确的谱系重建 (Nature Biotechnology 2022)。
Molecular recorders to track cellular events.
Deep distributed computing to reconstruct extremely large lineage trees.
Nature Biotechnology 40, 566-575, 2022 PubMed PDF
Retrospective clone isolation
遡及的クローン単離
回溯性克隆分离
Questions such as "Did the chemotherapy-resistant clones exist in the initial cell population with a unique cellular status from the beginning?" or "Did any molecular factor underlie the observed stem cell differentiation fates?" highlight many unresolved biological questions. These could be addressed if clones that have once demonstrated a specific phenotype in a later phase of cell progression can be isolated from the initial cell population. The new concept of “retrospective clone isolation” has recently emerged to tackle the above-raised questions. A barcoded cell population is first propagated in such a system, and its subpopulation is subjected to a given assay. After identifying a barcoded clone of interest, the same clone (or its close relative) is isolated in a barcode-specific manner from the initial or any other subpopulation stored during the experiment. The isolated live clone can then be subjected to any following experiments, including omics measurements and reconstitution of a synthetic cell population with the isolates. We have recently established a high-performance retrospective isolation technology CloneSelect using CRISPR base editing (bioRxiv 2022). We have demonstrated that CloneSelect is applicable in human cancer cell lines, human pluripotent stem cells, mouse stem cells, yeast cells, and E. coli cells.
「薬剤耐性を獲得して増殖するがん細胞のクローンは薬剤耐性獲得前の初期細胞集団に潜んでいたのでしょうか?」「幹細胞を分化させるときに、細胞ごとに分化のしやすさなど運命が決定付けられているのでしょうか?」このような疑問は、生物学における多くの未解決問題に根ざします。しかしながら、細胞集団を任意のアッセイで処理して特定の表現型を示したクローンを特定できた場合、これと同じクローンを初期細胞集団から単離するようなことができれば、これらの質問に答えることができるかもしれません。近年、遡及的クローン単離技術の開発が進んでいます。このアイディアでは、まず、集団内の多くの細胞をそれぞれ特異的な短いDNAバーコードでタグ付けします。これをわずかに増殖させたあと、その一部(サブポピュレーション)を目的のアッセイにかけます(幹細胞の細胞分化、オルガノイド構築、がん細胞の増殖実験など)。アッセイ後に特定の形質を示す細胞があるDNAバーコードをもっているクローンによって優勢的に占められていた場合は、これと同じクローンを、初期に保存されたサブポピュレーションからバーコード依存的に単離します。単離したクローンは、さまざまなオミクス計測や一度運命の特定されたクローンの複合による人工的な細胞社会の再構成などに使用することができます。私たちは最近、CRISPR塩基編集を利用した高感度の追溯的クローン単離手法CloneSelectを確立しました (bioRxiv 2022)。CloneSelectは、ヒトがん細胞、ヒト多能性幹細胞、マウス幹細胞、酵母細胞、および大腸菌細胞で利用可能です。
诸如"耐药癌细胞克隆是否从一开始就以独特的细胞状态潜伏于初始细胞群中?"或"干细胞的命运决定是否由某种分子或通路所调控?"等问题凸显了许多亟待解决的生物学谜团。如果我们能从初始细胞群中分离出在细胞增殖任意时期呈现出特殊表型的克隆,这些疑问将引刃而解。为实现这一设想,我们提出了"回溯性克隆分离"这个全新概念。首先,我们使用特定的DNA条形码分别标记初始细胞群中的每个细胞,并将它们用于细胞生长实验,例如干细胞分化、类器官培养、以及癌细胞增殖。在对细胞亚群进行观察和分析过程中,我们将确定感兴趣的细胞克隆,并识别它们所携带的DNA条形码。采用针对该条形码的报告系统,我们将从初始细胞群或实验期间储存的任何细胞亚群中提取出相同的克隆或其近亲。分离出的克隆可用于多种下游实验,包括多组学分析、以及通过结合特定命运的克隆进行细胞种群的合成重组。我们最近利用CRISPR碱基编辑技术建立了一种高性能追溯性克隆分离技术 CloneSelect (bioRxiv 2022)。实验证明,CloneSelect适用于各种人类癌症细胞系和多能干细胞、小鼠干细胞、酵母细胞、及大肠杆菌。
A multi-kingdom genetic barcoding system for precise target clone isolation
Automation of Laboratory and Data Analysis
生命科学実験とデータ解析の自動化
生命科学实验和数据分析的自动化
We have also been contributing to the development of laboratory automation and data analysis automation platforms. We have previously established humanoid robots to automate various molecular and cellular biology experiments and proposed the vision of robotic cloud biology (Nature Biotechnology 2017). More recently, we have established a new semantic framework to efficiently share reproducible DNA materials and construction protocols within the broad life sciences community (Nature Communications 2022). Furthermore, we have developed a universal tool to interpret and translate high-throughput sequencing read structures, thereby accelerating the development of new sequencing-based assays (Science Advances 2023). In the genomics space, we have established a new theoretical framework to scalably measure the spatial genomics of large tissues without the need for a microscopy system (Cell Systems 2023).
私たちはさらに、私たちの研究ビジョンを加速して実現させるために、生命科学実験およびデータ解析の自動化プラットフォームの開発に取り組んでいます。これまでにさまざまな分子および細胞生物学の実験を汎用的に自動化するために、ヒューマノイドを利用したRobotic Crowd Biology (Nature Biotechnology 2017)、広範なライフサイエンスコミュニティ内で再現可能なDNA試料およびDNA構築プロトコルを共有するセマンティックフレームワーク (Nature Communications 2022) を提案しています。さらに、次世代DNAシークエンシングリードの構造を汎用的に解釈して様々なソフトウェアのために翻訳するためのツール (Science Advances 2023) を開発しました。空間ゲノミクスの分野では、顕微鏡システムに依存しない大きな組織の空間ゲノミクスを計測する新しい理論的枠組みを開発しています (Cell Systems 2023)。
我们还致力于自动化生命科学实验和数据分析平台的开发,以加快实现我们的研究构想。此前,我们开发了用于自动化各种分子和细胞生物学实验的仿生机器人,并提出了机器人云生物学的概念 (Nature Biotechnology 2017)。最近,我们建立了能够在广泛科研社群中高效分享可重现DNA构建方案的新型语义框架 (Nature Communications 2022),及用于解码高通量测序读数结构以便下游生物学分析的通用工具 (Science Advances 2023)。另外,在空间转录组学领域,我们还开发了一个无需显微镜系统便可绘制大型组织样本中基因表达模式的理论框架 (Cell Systems 2023)。
Robotic crowd biology with Maholo LabDroids.
Nature Biotechnology 35, 310-312, 2017 PubMed PDF
A framework to efficiently describe and share reproducible DNA materials and construction protocols.
Nature Communications 13, 2894, 2022 PubMed PDF
A universal sequencing read interpreter.
Science Advances 9, eadd2793, 2023 PubMed PDF
DNA-GPS: A theoretical framework for optics-free spatial genomics and synthesis of current methods.
Cell Systems 14, 844-859.e4, 2023 PubMed PDF
Positions Available
メンバー募集
人才募集
We are seeking creative and talented postdocs and students.
Please see here if you are interested in joining us.
当研究室では現在、リスクをとって多分野の技術を組合わせたバイオテクノロジーを一緒に開発するメンバー、学生を募集しています。詳細はこちらをご覧下さい。
本研究室招募海内外优秀学生及博士后,共同开发多学科交叉、跨领域的革新生物技术。详情请参阅此处。









